Le variant Omicron apparaît mieux équipé pour reconnaître le récepteur cellulaire ACE2 de la souris que celui de l’homme, rapportent des chercheurs américains dans une étude publiée le 18 octobre 2022 dans les Comptes rendus de l’Académie américaine des sciences (PNAS). On rappelle que le coronavirus SARS-CoV-2 se fixe par l’intermédiaire de la région RBD (receptor binding domain, domaine de liaison au récepteur) de sa protéine spike au récepteur cellulaire ACE2 qui lui sert de porte d’entrée dans les cellules qu’il infecte.
On sait que le virus SARS-CoV-2 a une origine zoonotique : il a pour ancêtre un virus circulant chez des chauves-souris. Une fois que ce coronavirus a franchi la barrière d’espèce pour se transmettre chez l’homme, le domaine RBD de sa protéine spike a accumulé les mutations, dont certaines participent à sa capacité d’échapper à l’action du système immunitaire humain. Aujourd’hui, le variant Omicron du SARS-CoV-2 représente la souche dominante.
Si l’on considère le sous-variant Omicron BA.2, sa protéine spike diffère de celle présente dans la souche ancestrale. Plus précisément, le RBD de ce sous-variant Omicron diffère de celui de la souche ancestrale par 16 acides aminés, dont 7 se situent dans la sous-région (RBM, receptor binding motif, motif de liaison au récepteur, lui-même situé dans le RBD), entrant directement en contact avec le récepteur cellulaire ACE2. Le variant Omicron renferme un nombre significativement plus important de mutations que les variants qui l’ont précédé dans l’histoire évolutive de ce virus pandémique. Cela amène à se poser la question de l’origine de ce variant porteur d’une telle constellation de mutations.
Parmi les hypothèses, on a évoqué la possibilité que ce variant ait pu émerger dans un organisme humain qui aurait hébergé le SARS-CoV-2 pendant une très longue durée. Au sein d’un individu immunodéprimé, l’infection persistante par le SARS-CoV-2 aurait ainsi pu favoriser l’émergence d’un virus génétiquement distinct du virus qui l’avait infecté au départ et qui aurait acquis au fil du temps une multitude de mutations.
Une autre hypothèse postule que l’origine du variant Omicron soit animale et que la constellation de mutations dont il est porteur soit la résultante d’une adaptation préalable chez un animal. En d’autres termes, Omicron aurait pu infecter un hôte animal, au sein duquel il aurait acquis une masse critique de mutations, avant de franchir la barrière d’espèce dans l’autre sens pour se transmettre à l’homme.
Pour tenter de répondre à cette question, Wei Zhang, Hideki Aihara, Fang Li et leurs collègues de la faculté de médecine du Minnesota à Minneapolis ont comparé, sur le plan biochimique et structural, la capacité du RBD du variant Omicron BA.2 à reconnaître le récepteur ACE2 présent à la fois à la surface des cellules humaines et de souris.
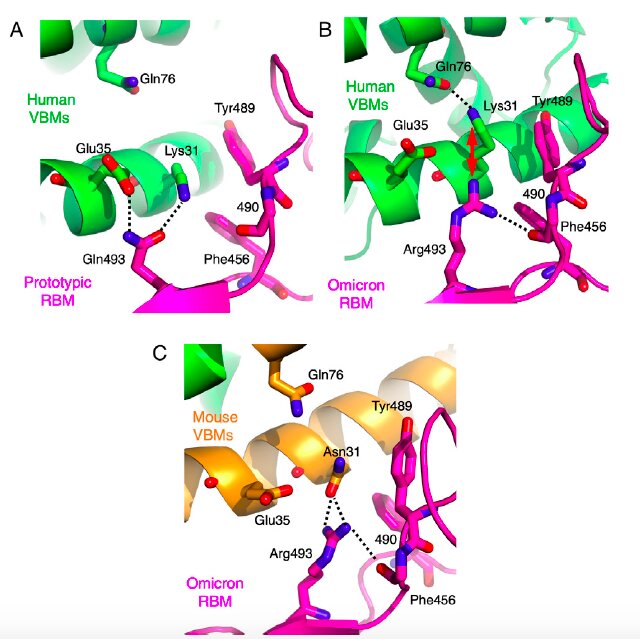
Les biochimistes et biologistes moléculaires se sont intéressés à quatre mutations situées dans le RBD du variant Omicron (Q493R, Q498R, N501Y, Y505H). Elles siègent à des emplacements connus pour être les sites privilégiés de l’apparition de mutations de la protéine spike, ce que l’on appelle des hotspots. Elles sont en regard de deux positions sur le récepteur humain ACE2 : l’une centrée sur l’acide aminé lysine en position 31 (Lys31), alors que l’autre concerne la lysine en position 353 (Lys353).
Les chercheurs américains ont tout d’abord montré que la mutation N501Y (localisée autour du hotspot 353) augmente significativement l’affinité du RBD d’Omicron pour le récepteur humain ACE2 et, à l’inverse, que les mutations Q498R (localisée autour du hotspot 31) et Y505H (localisée autour du hotspot 353) n’ont pas d’impact significatif sur la capacité du RBD à reconnaître le récepteur ACE2 humain. Au total, le RBD d’Omicron se lie plus efficacement au récepteur humain ACE2 que ne le fait le RBD de la souche ancestrale du SARS-CoV-2.
La capacité avec laquelle le RBD d’Omicron se lie au récepteur ACE2 de la souris a également été évaluée par les chercheurs. Leurs résultats indiquent que les quatre mutations (Q493R, Q498R, N501Y, Y505H) situées autour des hotspots 31 et 353 contribuent à renforcer la liaison du RBD au récepteur murin ACE2.
Afin de comprendre l’impact de ces quatre mutations du RBD d’Omicron sur sa capacité de liaison avec le récepteur ACE2 de la souris, les chercheurs ont déterminé par cristallographie aux rayons X la structure de l’interface entre le RBD et le récepteur murin ACE2 et celle formée entre le RBD et le récepteur humain ACE2.
Leurs analyses ont d’abord porté sur l’interface entre le hotspot 31 du RBD et le récepteur humain ACE2. Elles ont montré que la mutation Q493R déstabilise l’interface entre le RBD et le récepteur humain ACE2. En revanche, cette même mutation Q493R est compatible avec la présence de l’acide aminé asparagine (Asn31) dans le récepteur murin ACE2.
Les chercheurs se sont ensuite intéressés aux trois mutations centrées sur le hotspot 353 et à leur impact respectif sur le plan structural avec le récepteur humain ACE2. Il s’avère que la mutation N501Y est structurellement adaptée à la Lys353, que la mutation Y505H est incompatible avec la Lys353 et que la mutation Q498R est également incompatible avec le récepteur humain ACE2, mais que cette dernière est structurellement adaptée au récepteur murin ACE2.
En regard du hotspot 353 du RBD de la protéine spike virus, le récepteur cellulaire ACE2 diffère significativement entre l’homme et la souris. Dans le récepteur ACE2 murin, en position 353, l’acide aminé histidine (His353) remplace la lysine (Lys353) présente dans le récepteur ACE2 humain. Ceci explique que la souche ancestrale du SARS-CoV-2 ne se lie pas au récepteur murin ACE2 et qu’elle n’infecte que faiblement la souris.
Contrairement à la souche ancestrale, Omicron infecte efficacement la souris
Rien de tel avec le variant Omicron dont les mutations du RBD au niveau du hotspot 353 concourent à l’inverse à renforcer la liaison avec le récepteur murin ACE2, ce qui explique que ce variant infecte efficacement la souris.
De fait, les résultats des analyses montrent que les trois mutations Q498R, N501Y, Y505H, centrées sur le hotspot 353, sont structurellement adaptées à l’His353 présente dans le récepteur murin ACE2 (qui, répétons-le, a remplacé Lys353 présente dans le récepteur humain).
Les analyses de la liaison entre le RBD de la protéine spike d’Omicron et le récepteur ACE2 révèlent donc que les mutations présentes dans ce variant sont structurellement adaptées au récepteur ACE2 de la souris, bien plus qu’elles ne le sont du récepteur ACE2 humain.
La présence de ces mutations ne semble donc pas résulter d’une adaptation du virus pour le récepteur ACE2 humain. En revanche, ces mêmes mutations contribuent à augmenter l’affinité du RBD d’Omicron pour le récepteur ACE2 de la souris.
Trois mutations spécifiquement adaptées au récepteur murin ACE2
Ainsi, l’ensemble des données biochimiques et structurales obtenues montre que les trois mutations Q498R, N501Y, Y505H sont « spécifiquement adaptées » au récepteur ACE2 de la souris. Globalement, « le RBD d’Omicron est significativement mieux adapté au récepteur ACE2 murin qu’au récepteur ACE2 humain ».
Dans le récepteur ACE2 murin, on observe en position 31 une asparagine (Asn31) et en position 353 une histidine (His353), alors que ces deux emplacements sont occupés par une lysine dans le récepteur ACE2 humain.
Il se trouve par ailleurs que la souris est la seule espèce animale dont le récepteur ACE2 renferme Asn31 et His353, ce qui suggère qu’Omicron a évolué chez la souris avant de se diffuser chez l’homme et que les trois mutations présentes dans son RDB sont les traces laissées sur le variant Omicron lors de son évolution dans cette espèce murine.
Les données présentées par les scientifiques montrent que ces quatre mutations sont sans doute la conséquence de l’évolution du SARS-CoV2 chez la souris qui lui a permis de s’adapter au mieux au récepteur ACE2 murin.
Il s’avère que, parmi les quatre mutations, trois d’entre elles (Q493R, Q498R, Y505H) sont « adaptées de manière unique » à la reconnaissance du récepteur ACE2 de la souris, alors que la mutation N501Y est adaptée à la fois au récepteur ACE2 humain et murin. Il s’avère que ces mutations Q493R, Q498R, N501Y, Y505H permettent au RBD d’Omicron de mieux reconnaître le récepteur ACE2 murin que le récepteur ACE2 humain.
Omicron était bien adapté au récepteur ACE2 de la souris avant de commencer à infecter l’homme
Ces données apportent donc un nouvel éclairage sur l’évolution d’Omicron. Elles suggèrent que la souris a été l’animal dans lequel le variant Omicron a émergé.
Selon les auteurs de l’étude, un variant du SARS-CoV-2 renfermant la mutation N501Y (structurellement adaptée à plus d’une espèce animale) aurait pu être transmis à partir de l’homme (ou d’un animal) à la souris. Après quoi, le coronavirus aurait évolué dans cette espèce murine, aurait progressivement acquis des mutations spécifiques de la souris dans le motif de liaison au récepteur, RBM, de la protéine spike, telles que les mutations Q493R, Q498R et Y505H. Tout ceci aurait contribué à l’émergence, chez la souris, du variant Omicron.
Ce n’est pas la première fois que la piste animale du variant Omicron est évoquée. En novembre 2021, des chercheurs de l’Académie chinoise des sciences ont rapporté, dans le Journal of Genetics and Genomics (revue éditée à Pékin en langue anglaise) des résultats allant dans le sens d’une évolution prolongée du SARS-CoV-2 au sein de l’espèce murine avant qu’il ne migre à nouveau chez l’homme (spillback).
Ces données n’ont cependant pas suffi à convaincre l’équipe de Jesse Bloom (université de Chicago), qui étudie l’évolution moléculaire des virus. Ces chercheurs considèrent toujours que les caractéristiques évolutives inhabituelles d’Omicron s’expliquent mieux par des infections humaines chroniques, autrement dit que ce variant a émergé chez des immunodéprimés.
Nécessité d’une surveillance épidémiologique chez les rongeurs
Selon les chercheurs américains, en plus d’infecter l’espèce humaine, le variant Omicron peut se transmettre à d’autres espèces animales dont le récepteur ACE2 renferme des acides aminés pouvant être reconnus par le RBD de la protéine spike d’Omicron. À ce propos, ils font remarquer que les récepteurs ACE2 de certaines espèces de rat contiennent soit Asn31 (asparagine en position 31), soit His353 (histidine en position 353). Et de conclure qu’ « une surveillance épidémiologique renforcée chez les souris et les rats devraient aider à clarifier l’évolution et la transmission du variant Omicron ».
Une équipe de chercheurs français ne dit pas autre chose dans un article paru le 13 octobre sur le site de prépublication bioRxiv, dans lequel ils rapportent que deux rats domestiques ont été contaminés par leur propriétaire infecté par le SARS-CoV-2. L’équipe, dirigée par Étienne Simon-Lorière de l’Institut Pasteur (Paris), estime que les rongeurs devraient faire l’objet d’une surveillance épidémiologique au niveau de l’interface homme-animal, aussi bien dans les grandes villes que dans les zones rurales.
Marc Gozlan (Suivez-moi sur Twitter, Facebook, LinkedIn, et sur mon autre blog ‘Le diabète dans tous ses états’, consacré aux mille et une facettes du diabète – déjà vingt-cinq billets.)
Pour en savoir plus :
Zhang W, Shi K, Geng O, et al. Structural basis for mouse receptor recognition by SARS-CoV-2 omicron variant. Proc Natl Acad Sci U S A. October 18, 2022. doi: 10.1073/pnas.2206509119
Montagutelli X, Decaudin B, Beretta M, et al. SARS-CoV-2 infection in domestic rats after transmission from their infected owner. bioRxiv. Posted October 13, 2022. doi: 10.1101/2022.10.13.512053
Wei C, Shan KJ, Wang W, et al. Evidence for a mouse origin of the SARS-CoV-2 Omicron variant. J Genet Genomics. 2021 Dec;48(12):1111-1121. doi: 10.1016/j.jgg.2021.12.003
Wu K, Peng G, Wilken M, et al. Mechanisms of host receptor adaptation by severe acute respiratory syndrome coronavirus. J Biol Chem. 2012 Mar 16;287(12):8904-11. doi: 10.1074/jbc.M111.325803
Sun S, Gu H, Cao L, et al. Characterization and structural basis of a lethal mouse-adapted SARS-CoV-2. Nat Commun. 2021 Sep 27;12(1):5654. doi: 10.1038/s41467-021-25903-x
Shuai H, Chan JF, Yuen TT, et al. Emerging SARS-CoV-2 variants expand species tropism to murines. EBioMedicine. 2021 Nov;73:103643. doi: 10.1016/j.ebiom.2021.103643
Huang K, Zhang Y, Hui X, et al. Q493K and Q498H substitutions in Spike promote adaptation of SARS-CoV-2 in mice. EBioMedicine. 2021 May;67:103381. doi: 10.1016/j.ebiom.2021.103381
Shang J, Ye G, Shi K, et al. Structural basis of receptor recognition by SARS-CoV-2. Nature. 2020 May;581(7807):221-224. doi: 10.1038/s41586-020-2179-y
Sur le web :
LIRE aussi : Covid-19 : des rats domestiques contaminés par leur propriétaire infecté par le SARS-CoV-2